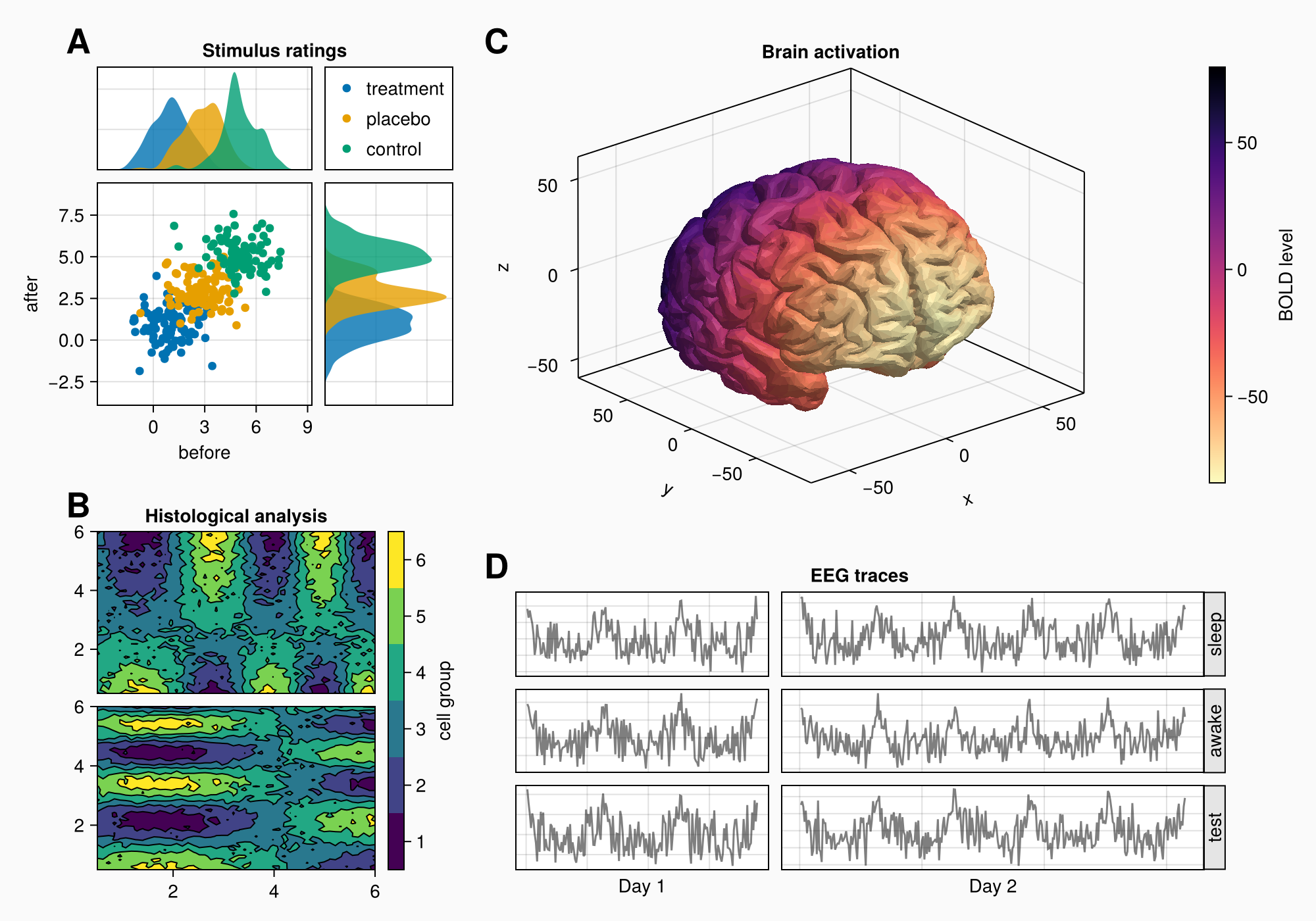
Создание сложных макетов
|
Страница в процессе перевода. |
В этом руководстве содержатся сведения о создании сложного рисунка с помощью инструментов построения макетов Makie.
Допустим, мы хотим создать следующий рисунок.
Далее приведен полный код для справки.
using CairoMakie
using Makie.FileIO
CairoMakie.activate!() # hide
f = Figure(backgroundcolor = RGBf(0.98, 0.98, 0.98),
size = (1000, 700))
ga = f[1, 1] = GridLayout()
gb = f[2, 1] = GridLayout()
gcd = f[1:2, 2] = GridLayout()
gc = gcd[1, 1] = GridLayout()
gd = gcd[2, 1] = GridLayout()
axtop = Axis(ga[1, 1])
axmain = Axis(ga[2, 1], xlabel = "before", ylabel = "after")
axright = Axis(ga[2, 2])
linkyaxes!(axmain, axright)
linkxaxes!(axmain, axtop)
labels = ["treatment", "placebo", "control"]
data = randn(3, 100, 2) .+ [1, 3, 5]
for (label, col) in zip(labels, eachslice(data, dims = 1))
scatter!(axmain, col, label = label)
density!(axtop, col[:, 1])
density!(axright, col[:, 2], direction = :y)
end
ylims!(axtop, low = 0)
xlims!(axright, low = 0)
axmain.xticks = 0:3:9
axtop.xticks = 0:3:9
leg = Legend(ga[1, 2], axmain)
hidedecorations!(axtop, grid = false)
hidedecorations!(axright, grid = false)
leg.tellheight = true
colgap!(ga, 10)
rowgap!(ga, 10)
Label(ga[1, 1:2, Top()], "Stimulus ratings", valign = :bottom,
font = :bold,
padding = (0, 0, 5, 0))
xs = LinRange(0.5, 6, 50)
ys = LinRange(0.5, 6, 50)
data1 = [sin(x^1.5) * cos(y^0.5) for x in xs, y in ys] .+ 0.1 .* randn.()
data2 = [sin(x^0.8) * cos(y^1.5) for x in xs, y in ys] .+ 0.1 .* randn.()
ax1, hm = contourf(gb[1, 1], xs, ys, data1,
levels = 6)
ax1.title = "Histological analysis"
contour!(ax1, xs, ys, data1, levels = 5, color = :black)
hidexdecorations!(ax1)
ax2, hm2 = contourf(gb[2, 1], xs, ys, data2,
levels = 6)
contour!(ax2, xs, ys, data2, levels = 5, color = :black)
cb = Colorbar(gb[1:2, 2], hm, label = "cell group")
low, high = extrema(data1)
edges = range(low, high, length = 7)
centers = (edges[1:6] .+ edges[2:7]) .* 0.5
cb.ticks = (centers, string.(1:6))
cb.alignmode = Mixed(right = 0)
colgap!(gb, 10)
rowgap!(gb, 10)
brain = load(assetpath("brain.stl"))
ax3d = Axis3(gc[1, 1], title = "Brain activation")
m = mesh!(
ax3d,
brain,
color = [tri[1][2] for tri in brain for i in 1:3],
colormap = Reverse(:magma),
)
Colorbar(gc[1, 2], m, label = "BOLD level")
axs = [Axis(gd[row, col]) for row in 1:3, col in 1:2]
hidedecorations!.(axs, grid = false, label = false)
for row in 1:3, col in 1:2
xrange = col == 1 ? (0:0.1:6pi) : (0:0.1:10pi)
eeg = [sum(sin(pi * rand() + k * x) / k for k in 1:10)
for x in xrange] .+ 0.1 .* randn.()
lines!(axs[row, col], eeg, color = (:black, 0.5))
end
axs[3, 1].xlabel = "Day 1"
axs[3, 2].xlabel = "Day 2"
Label(gd[1, :, Top()], "EEG traces", valign = :bottom,
font = :bold,
padding = (0, 0, 5, 0))
rowgap!(gd, 10)
colgap!(gd, 10)
for (i, label) in enumerate(["sleep", "awake", "test"])
Box(gd[i, 3], color = :gray90)
Label(gd[i, 3], label, rotation = pi/2, tellheight = false)
end
colgap!(gd, 2, 0)
n_day_1 = length(0:0.1:6pi)
n_day_2 = length(0:0.1:10pi)
colsize!(gd, 1, Auto(n_day_1))
colsize!(gd, 2, Auto(n_day_2))
for (label, layout) in zip(["A", "B", "C", "D"], [ga, gb, gc, gd])
Label(layout[1, 1, TopLeft()], label,
fontsize = 26,
font = :bold,
padding = (0, 5, 5, 0),
halign = :right)
end
colsize!(f.layout, 1, Auto(0.5))
rowsize!(gcd, 1, Auto(1.5))
fКак подойти к решению этой задачи?
В следующих разделах мы рассмотрим этот процесс пошагово. Мы не всегда будем использовать максимально короткий синтаксис, так как главная цель — лучше понять логику и доступные варианты.
Базовый план макета
При построении рисунков всегда берутся в расчет прямоугольники. Мы хотим найти самые большие прямоугольники, заключающие значимые группы содержимого, а затем реализовать их либо с помощью GridLayout, либо поместив в них объекты содержимого.
Если взглянуть на целевой рисунок, можно представить по одному прямоугольнику вокруг каждой из помеченных областей A, B, C и D. Но A и C не находятся в одной строке, как и B и D. Это означает, что мы не используем блок GridLayout 2 x 2, а должны найти более творческий подход.
Можно сказать, что A и B находятся в одном столбце, а C и D — в другом. Мы можем получить разную высоту строк для обеих групп, создав один большой вложенный блок GridLayout внутри второго столбца, в который мы поместим C и D. Таким образом, строки столбца 2 будут отделены от столбца 1.
using CairoMakie
using FileIO
f = Figure(backgroundcolor = RGBf(0.98, 0.98, 0.98),
size = (1000, 700))
Настройка блоков GridLayout
Теперь создадим четыре вложенных блока GridLayout, которые будут содержать объекты A, B, C и D. Есть также макет, который содержит C и D, так что строки отделены от A и B. Пока мы ничего не видим, поскольку у нас нет видимого содержимого, но это ненадолго.
|
Note Необязательно сначала создавать отдельные блоки |
ga = f[1, 1] = GridLayout()
gb = f[2, 1] = GridLayout()
gcd = f[1:2, 2] = GridLayout()
gc = gcd[1, 1] = GridLayout()
gd = gcd[2, 1] = GridLayout()Панель A
Теперь можно размещать объекты на рисунке. Начнем с A.
Имеются три оси и условные обозначения. Сначала мы можем разместить оси, связать их соответствующим образом и построить на них первые данные.
axtop = Axis(ga[1, 1])
axmain = Axis(ga[2, 1], xlabel = "before", ylabel = "after")
axright = Axis(ga[2, 2])
linkyaxes!(axmain, axright)
linkxaxes!(axmain, axtop)
labels = ["treatment", "placebo", "control"]
data = randn(3, 100, 2) .+ [1, 3, 5]
for (label, col) in zip(labels, eachslice(data, dims = 1))
scatter!(axmain, col, label = label)
density!(axtop, col[:, 1])
density!(axright, col[:, 2], direction = :y)
end
f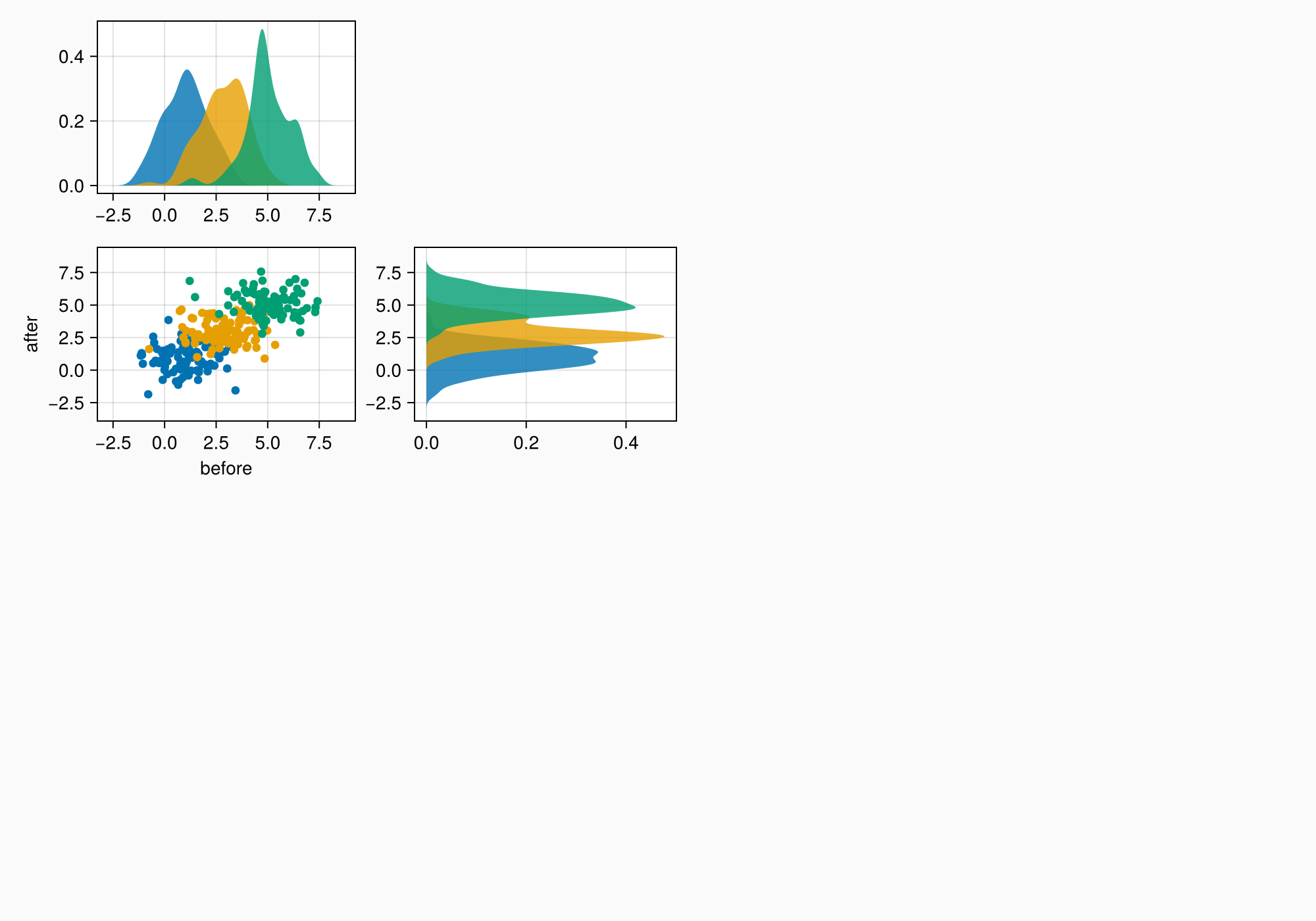
Между графиками плотности и их осями есть небольшой промежуток, который можно удалить, зафиксировав одну сторону пределов.
ylims!(axtop, low = 0)
xlims!(axright, low = 0)
f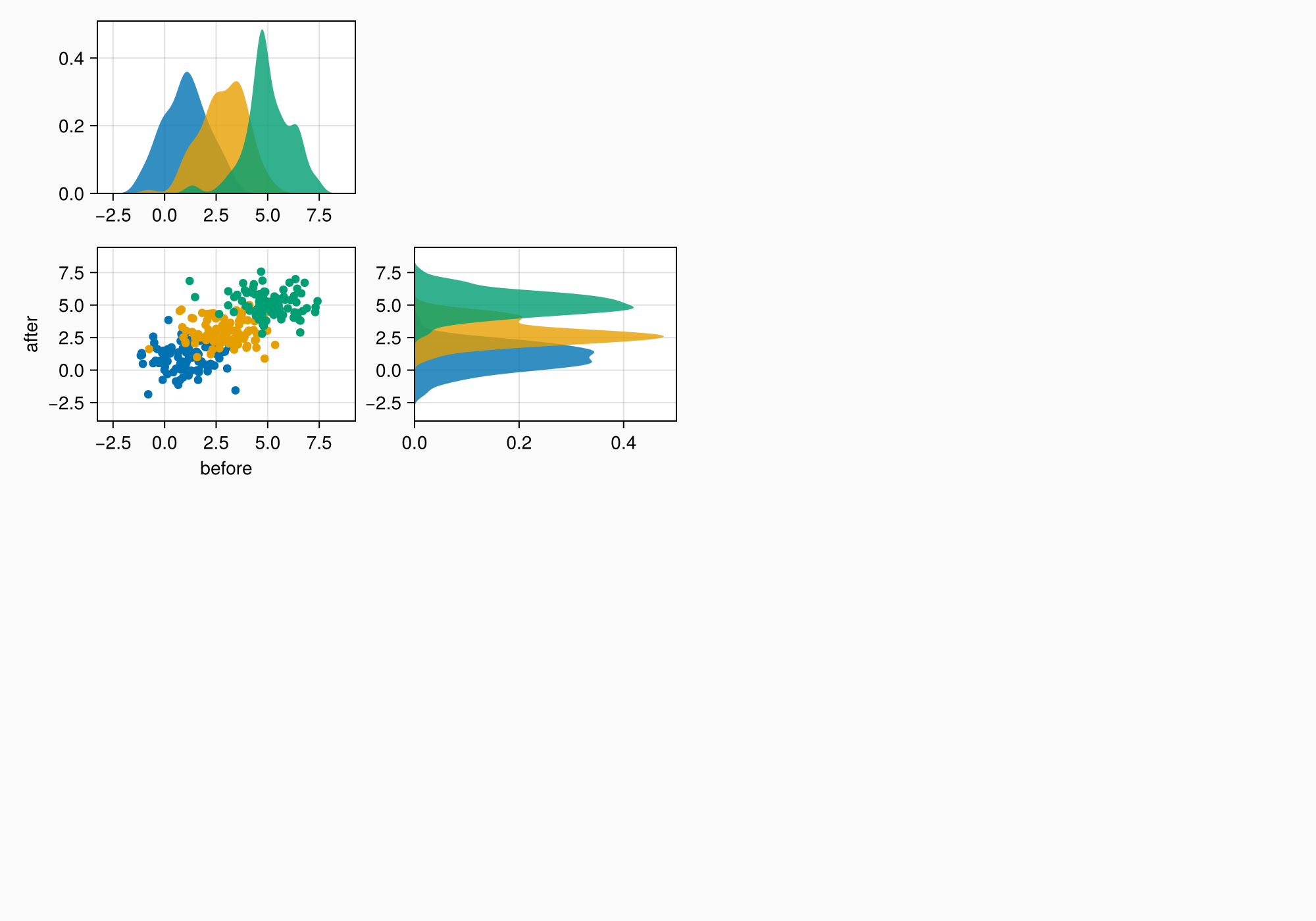
axmain.xticks = 0:3:9
axtop.xticks = 0:3:9
f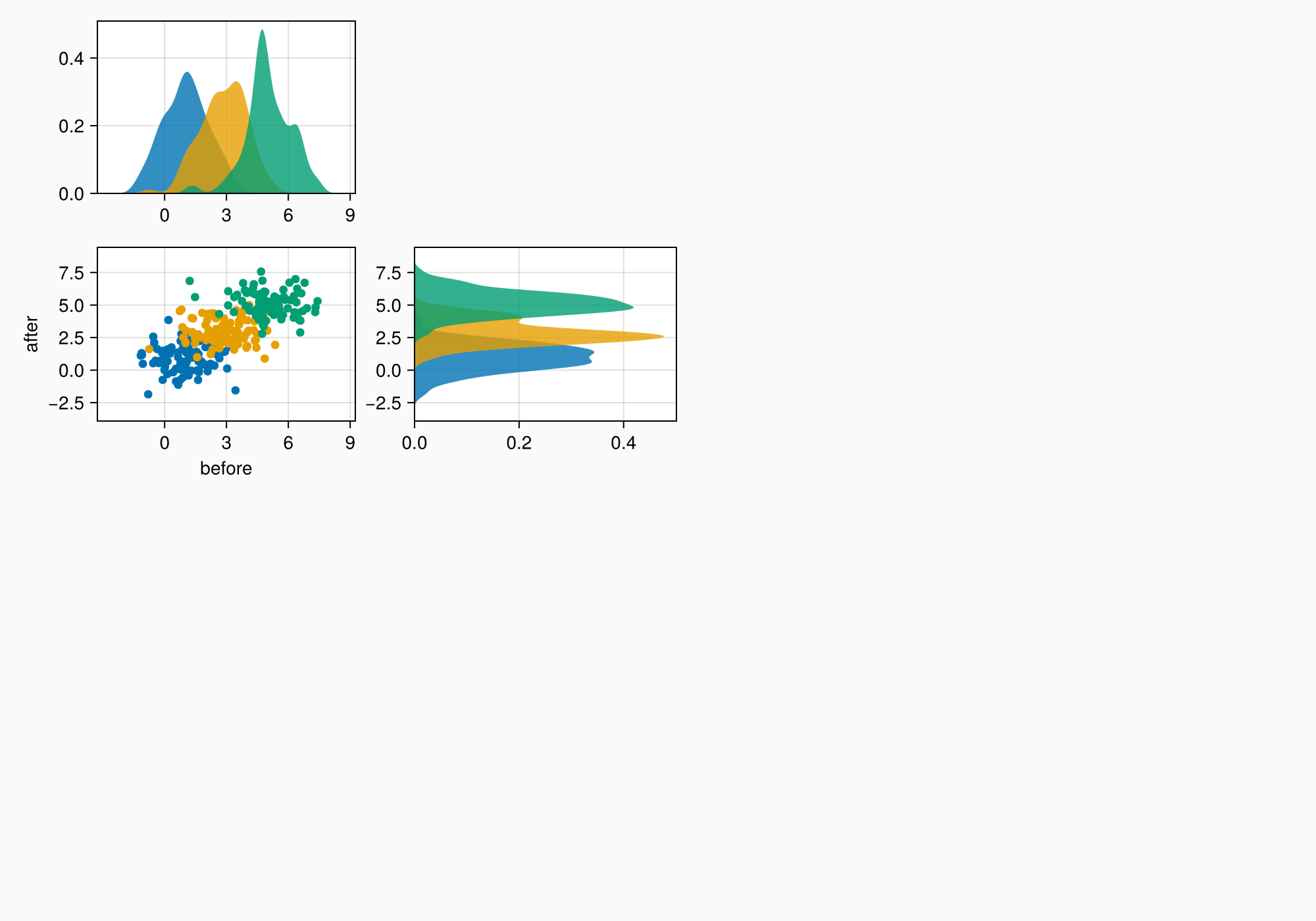
Условные обозначения
Мы задали атрибут label в вызове функции рассеяния, чтобы было проще создать условные обозначения. Можно просто передать axmain в виде второго аргумента в Legend.
leg = Legend(ga[1, 2], axmain)
f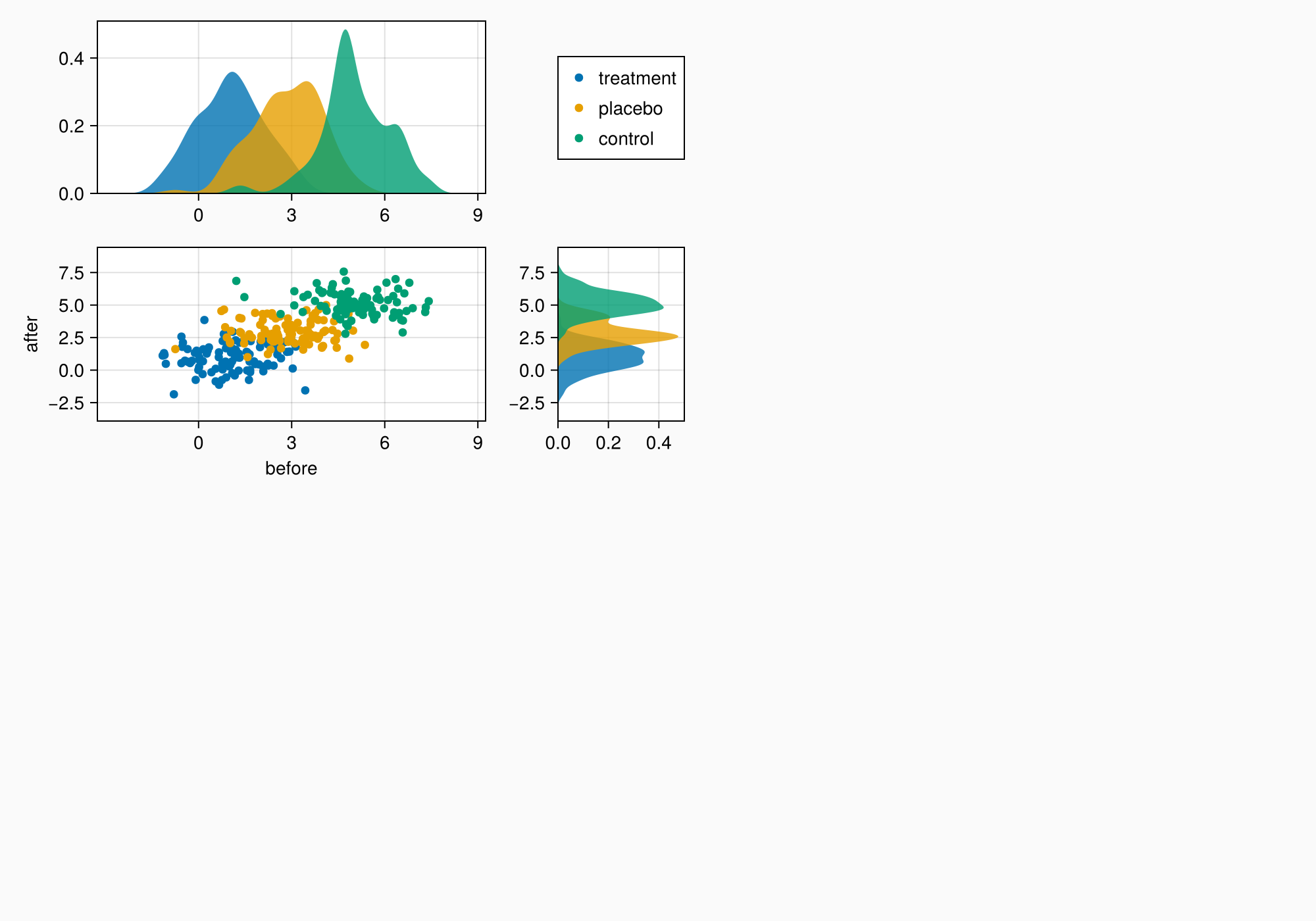
Настройки условных обозначений
Нам нужно изменить несколько моментов. У боковых осей есть ненужные дополнительные элементы, которые мы собираемся скрыть.
Кроме того, высота верхней оси отличается от высоты условных обозначений. Это связано с тем, что условные обозначения обычно используются справа от Axis и поэтому имеют предустановку tellheight = false. Зададим этому атрибуту значение true, чтобы строка, в которой находятся условные обозначения, могла сжиматься до известных размеров.
hidedecorations!(axtop, grid = false)
hidedecorations!(axright, grid = false)
leg.tellheight = true
f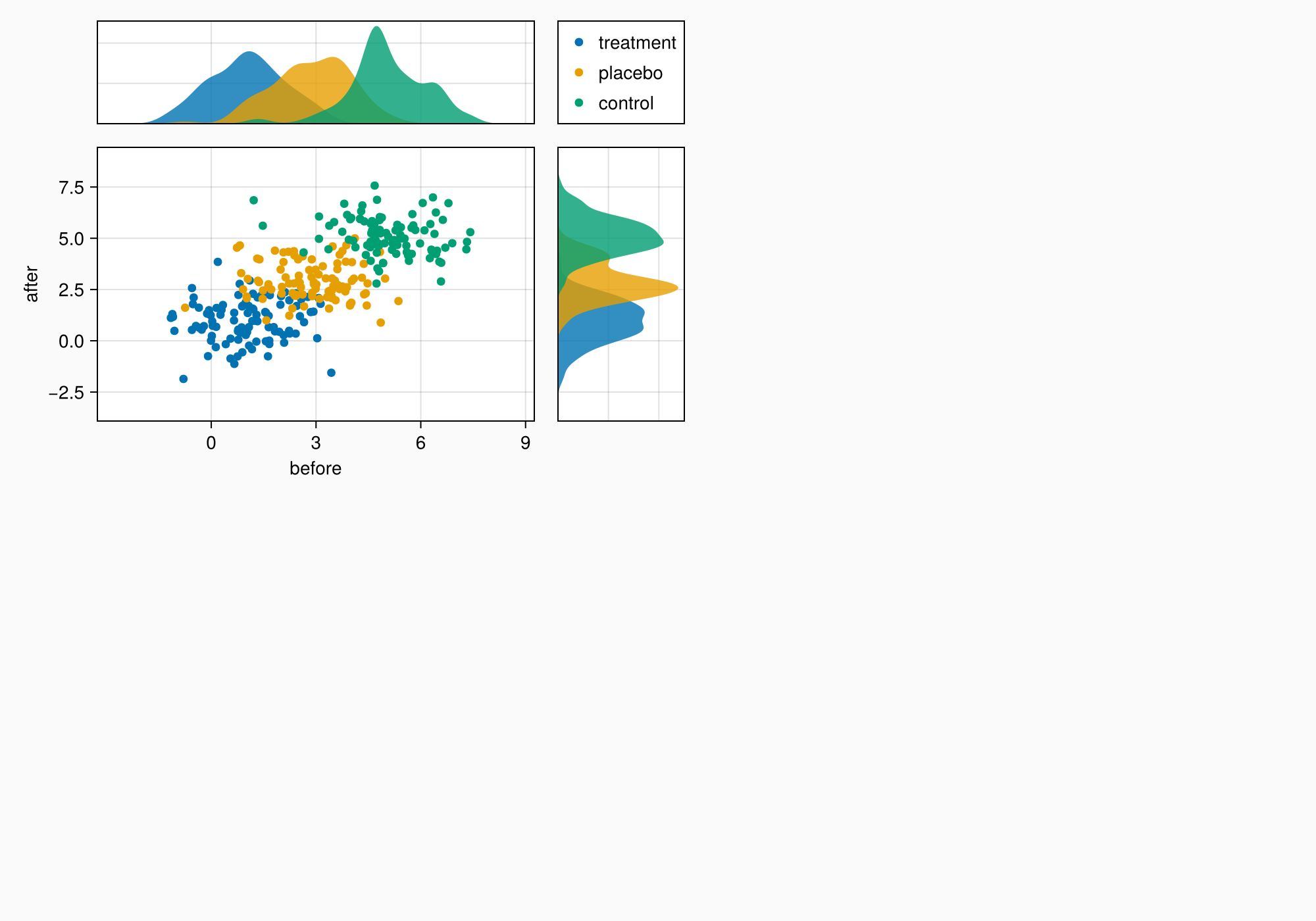
Оси все еще находятся слишком далеко друг от друга, поэтому сократим промежутки между столбцами и строками.
colgap!(ga, 10)
rowgap!(ga, 10)
f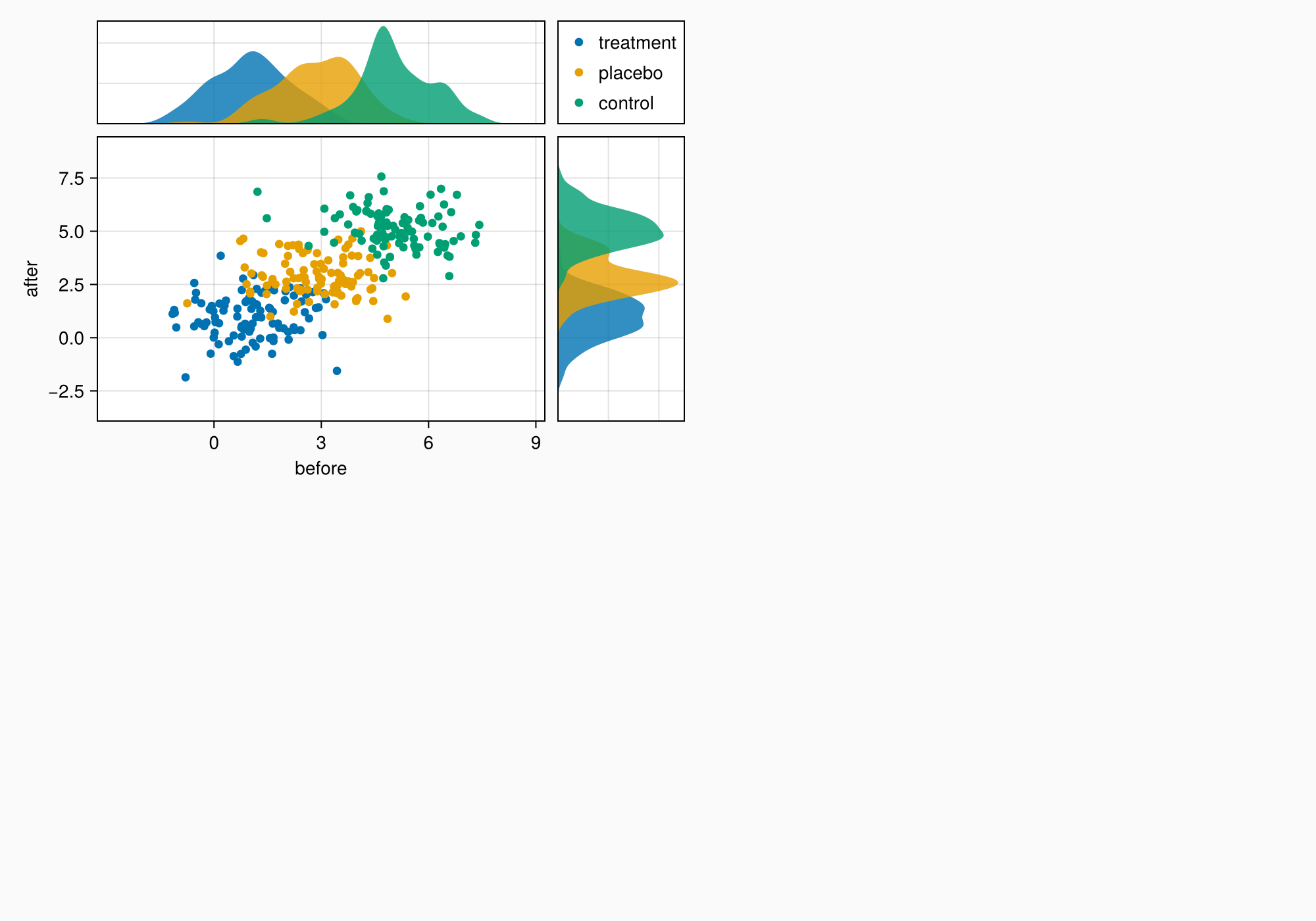
Label(ga[1, 1:2, Top()], "Stimulus ratings", valign = :bottom,
font = :bold,
padding = (0, 0, 5, 0))
f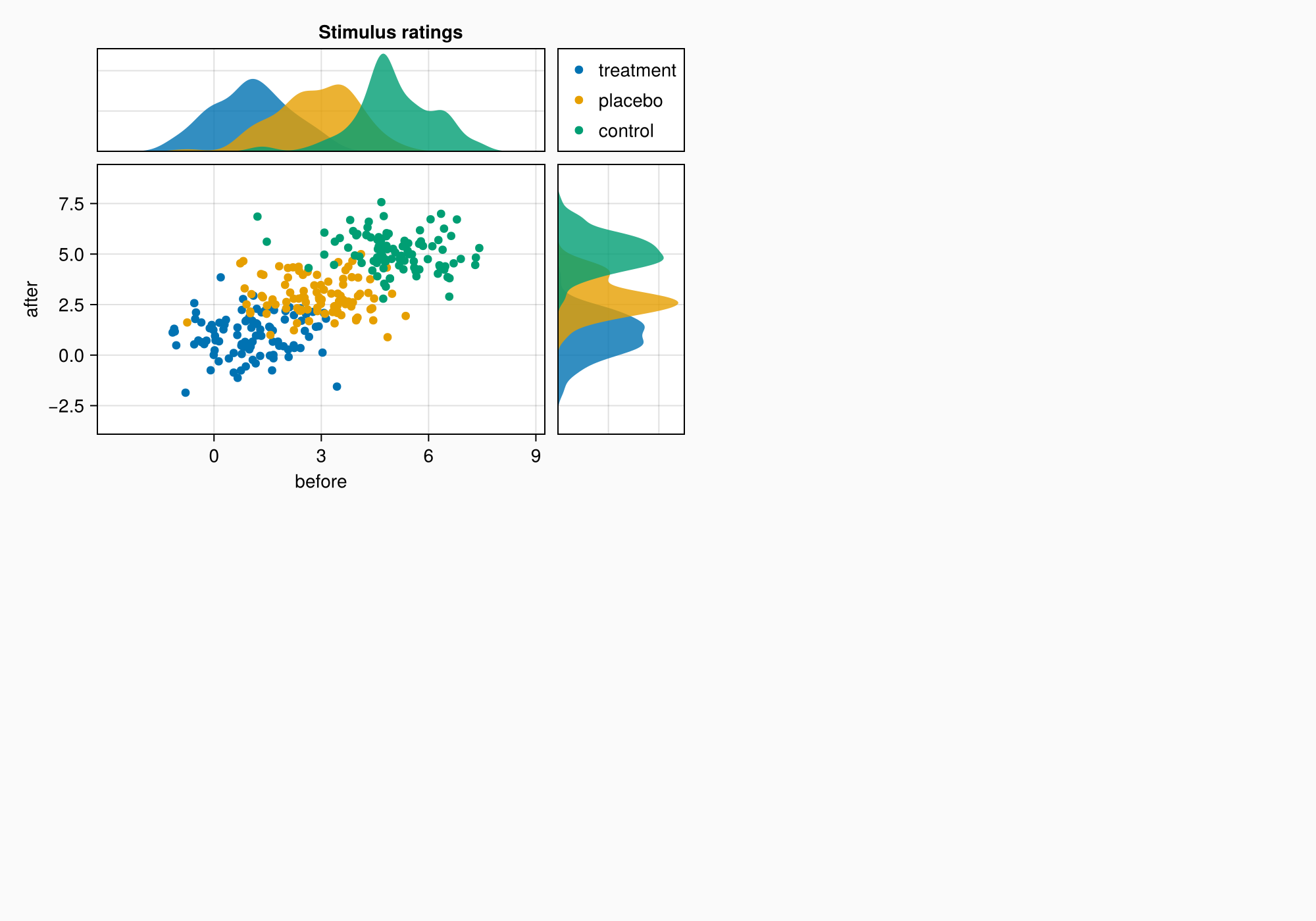
Панель B
Есть две оси, расположенные друг над другом, и панель цветов рядом с ними. На этот раз создадим оси, просто построив их в правые слоты GridLayout. Этот вариант удобнее изначального создания Axis.
xs = LinRange(0.5, 6, 50)
ys = LinRange(0.5, 6, 50)
data1 = [sin(x^1.5) * cos(y^0.5) for x in xs, y in ys] .+ 0.1 .* randn.()
data2 = [sin(x^0.8) * cos(y^1.5) for x in xs, y in ys] .+ 0.1 .* randn.()
ax1, hm = contourf(gb[1, 1], xs, ys, data1,
levels = 6)
ax1.title = "Histological analysis"
contour!(ax1, xs, ys, data1, levels = 5, color = :black)
hidexdecorations!(ax1)
ax2, hm2 = contourf(gb[2, 1], xs, ys, data2,
levels = 6)
contour!(ax2, xs, ys, data2, levels = 5, color = :black)
f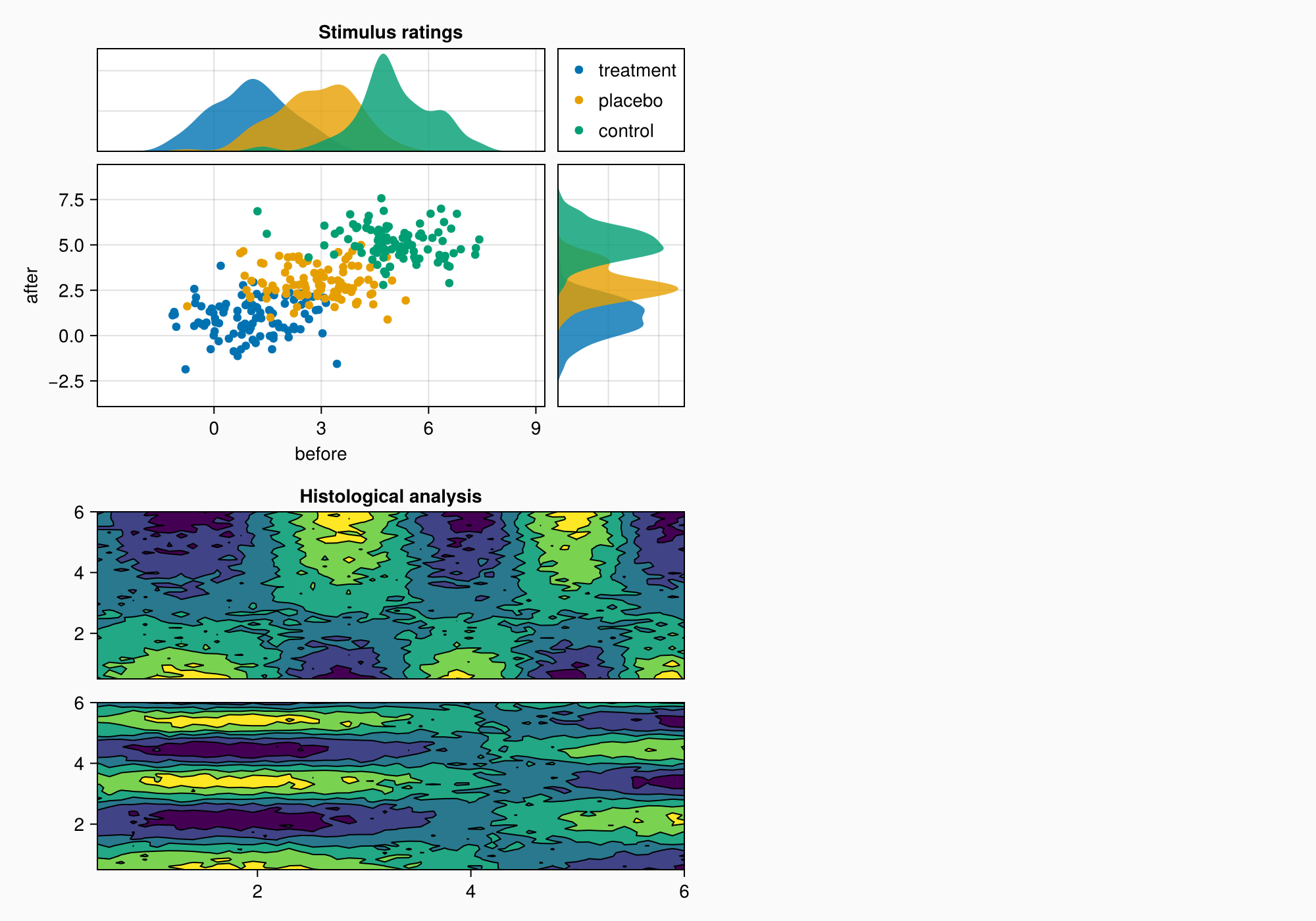
Панель цветов
Теперь нам нужна панель цветов Поскольку мы не задавали конкретных краев для двух контурных графиков, а только количество уровней, можно создать панель цветов, используя один из контурных графиков, а затем пометить столбцы от первого до шестого.
cb = Colorbar(gb[1:2, 2], hm, label = "cell group")
low, high = extrema(data1)
edges = range(low, high, length = 7)
centers = (edges[1:6] .+ edges[2:7]) .* 0.5
cb.ticks = (centers, string.(1:6))
f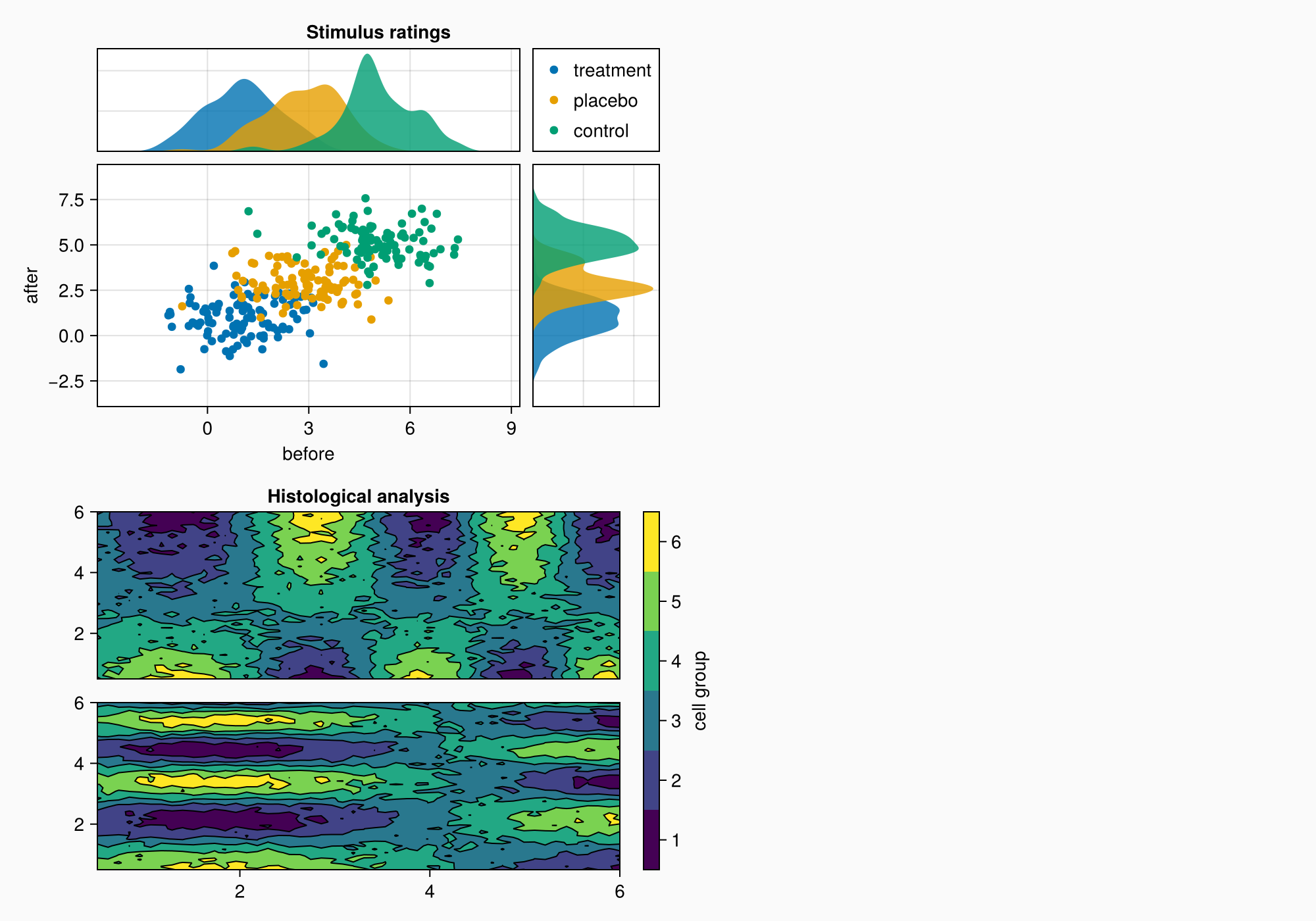
Смешанный режим выравнивания
Правый край панели цветов сейчас выровнен с правым краем верхнего графика плотности. Впоследствии из-за этого между графиком плотности и содержимым справа может образоваться промежуток.
Чтобы улучшить ситуацию, можно разместить метки панели цветов в ячейке макета, используя режим выравнивания Mixed. Ключевое слово right = 0 означает, что правая часть панели цветов должна включать внутри себя выступающее содержимое с дополнительным заполнением 0.
cb.alignmode = Mixed(right = 0)
f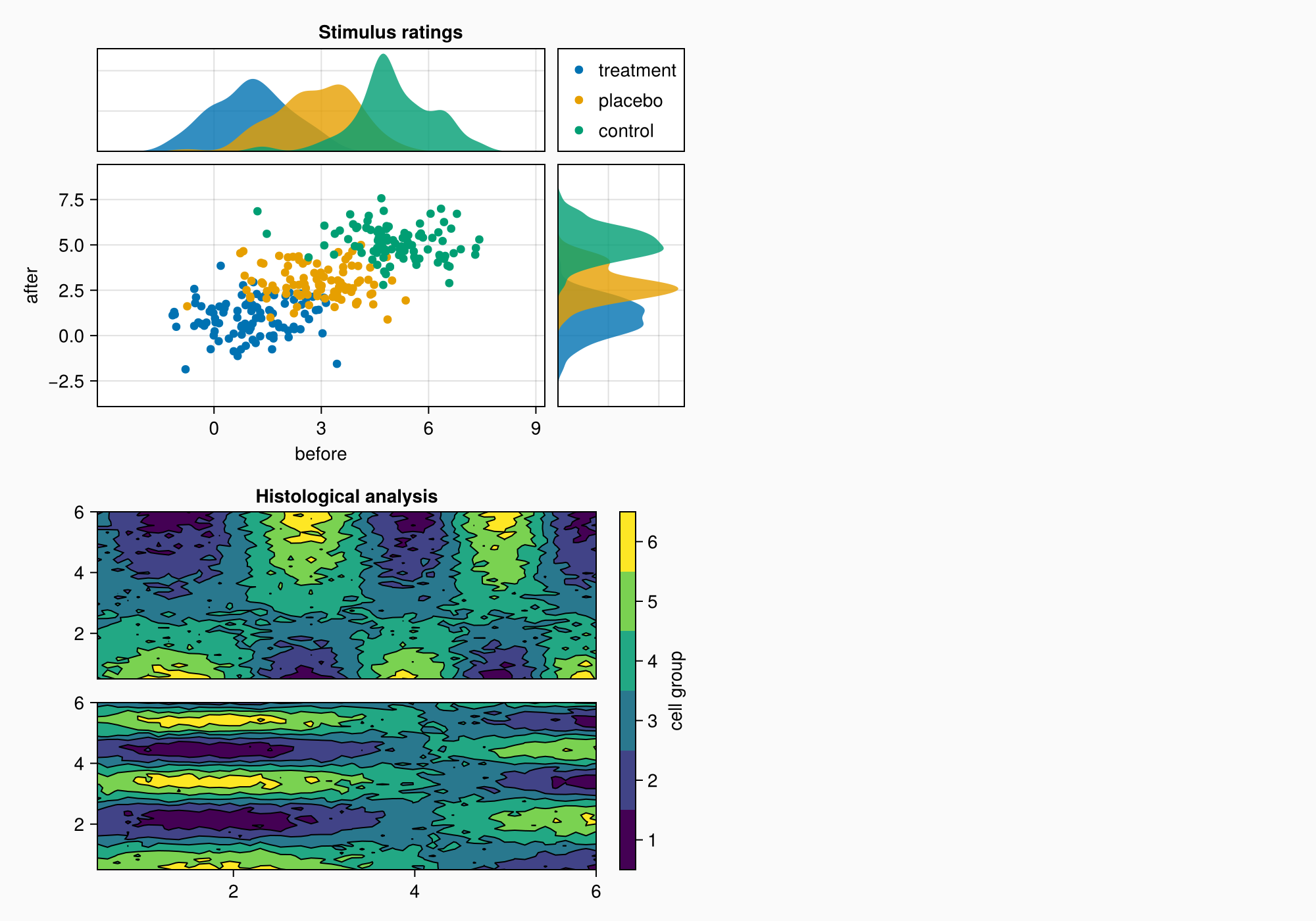
colgap!(gb, 10)
rowgap!(gb, 10)
f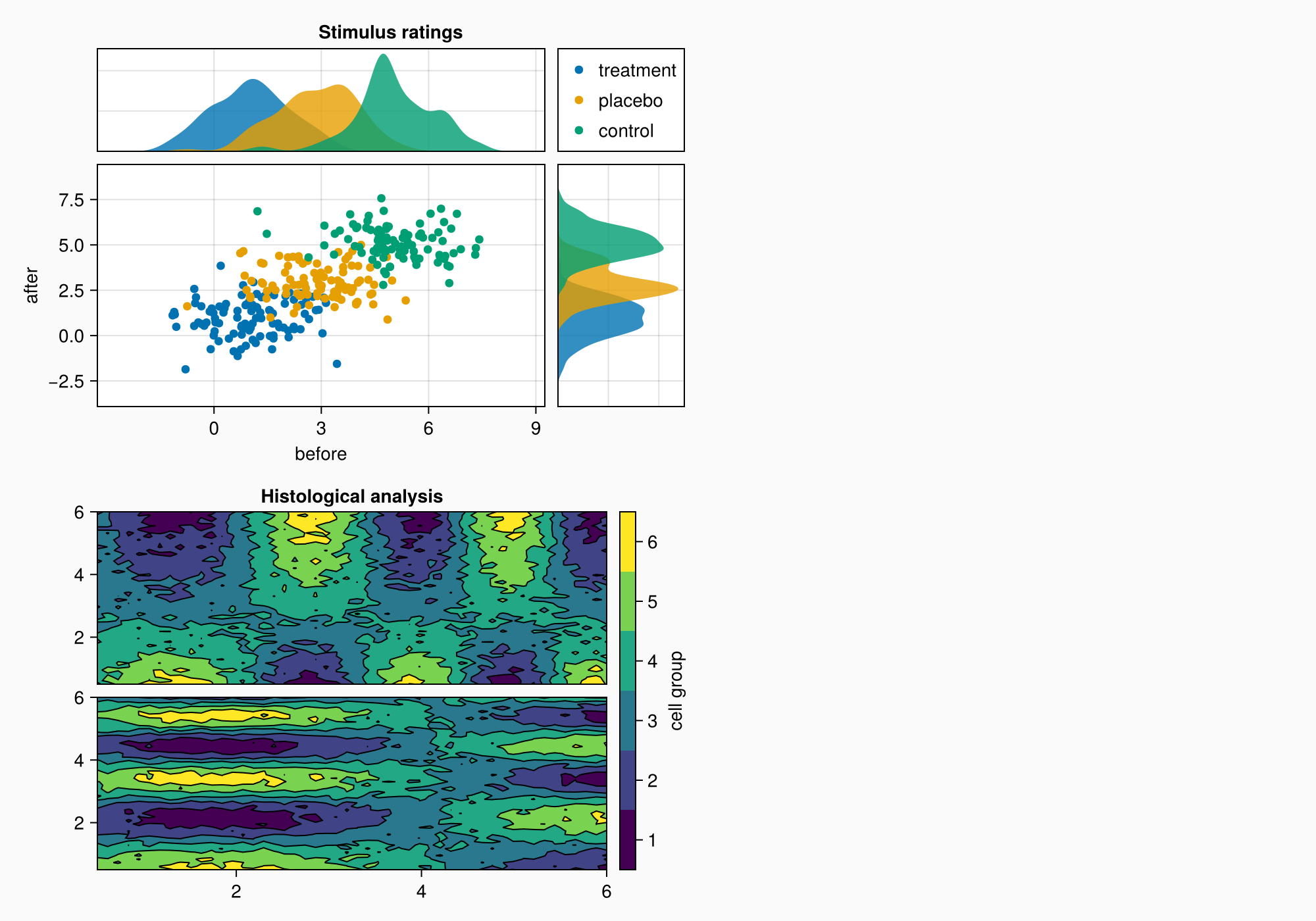
Панель C
brain = load(assetpath("brain.stl"))
ax3d = Axis3(gc[1, 1], title = "Brain activation")
m = mesh!(
ax3d,
brain,
color = [tri[1][2] for tri in brain for i in 1:3],
colormap = Reverse(:magma),
)
Colorbar(gc[1, 2], m, label = "BOLD level")
f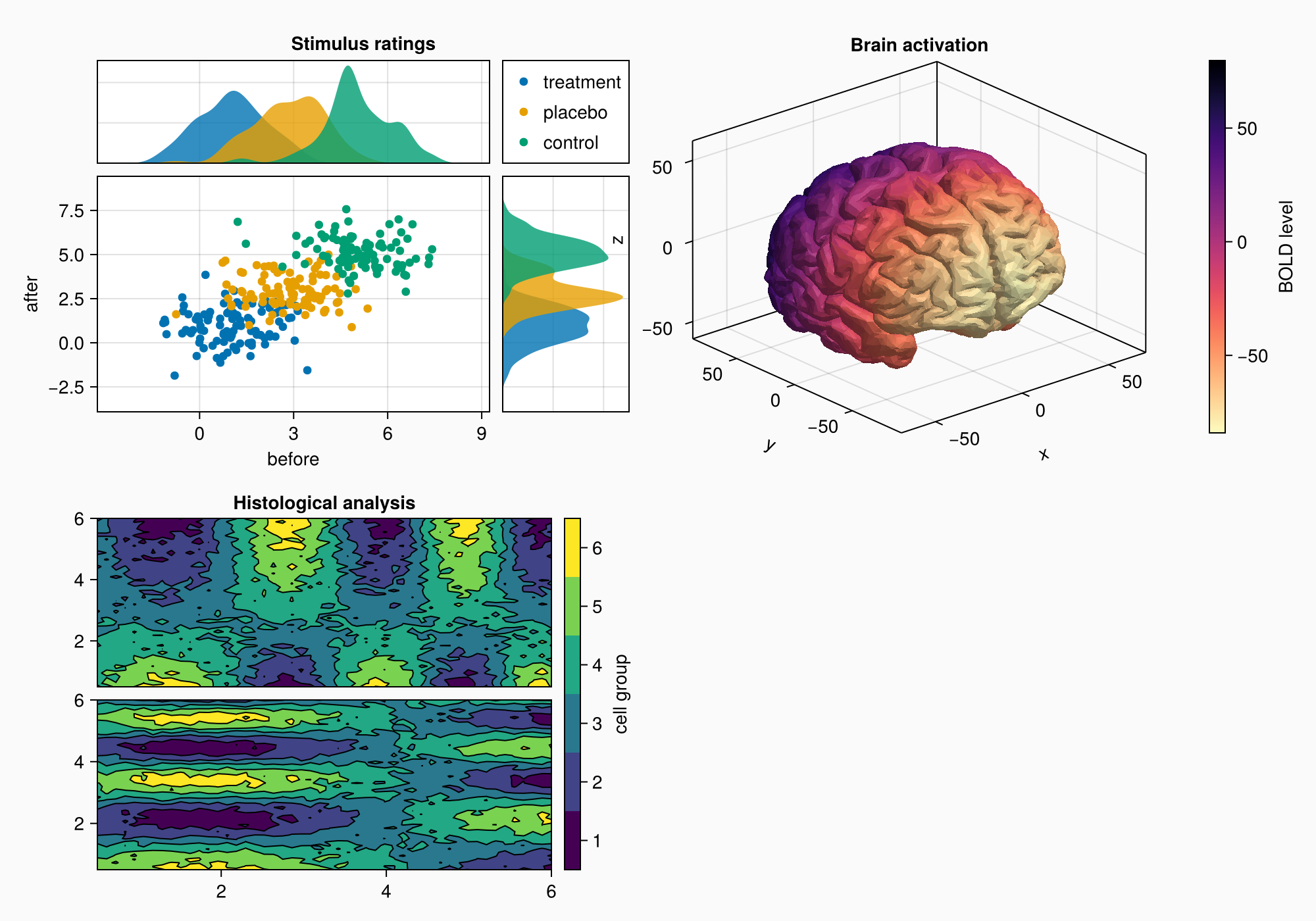
Обратите внимание, что метка z немного перекрывает график слева. У Axis3 не может быть автоматических выступов, потому что положение метки меняется в зависимости от проекции и размера ячейки оси — в этом отличие от двухмерного блока Axis.
Вы можете задать в качестве значения атрибута ax3.protrusions кортеж из четырех значений (лево, право, низ, верх), но в данном случае мы просто будем продолжать построение, пока не получим все нужные объекты, а затем посмотрим, нужны ли такие небольшие изменения.
Панель D
axs = [Axis(gd[row, col]) for row in 1:3, col in 1:2]
hidedecorations!.(axs, grid = false, label = false)
for row in 1:3, col in 1:2
xrange = col == 1 ? (0:0.1:6pi) : (0:0.1:10pi)
eeg = [sum(sin(pi * rand() + k * x) / k for k in 1:10)
for x in xrange] .+ 0.1 .* randn.()
lines!(axs[row, col], eeg, color = (:black, 0.5))
end
axs[3, 1].xlabel = "Day 1"
axs[3, 2].xlabel = "Day 2"
f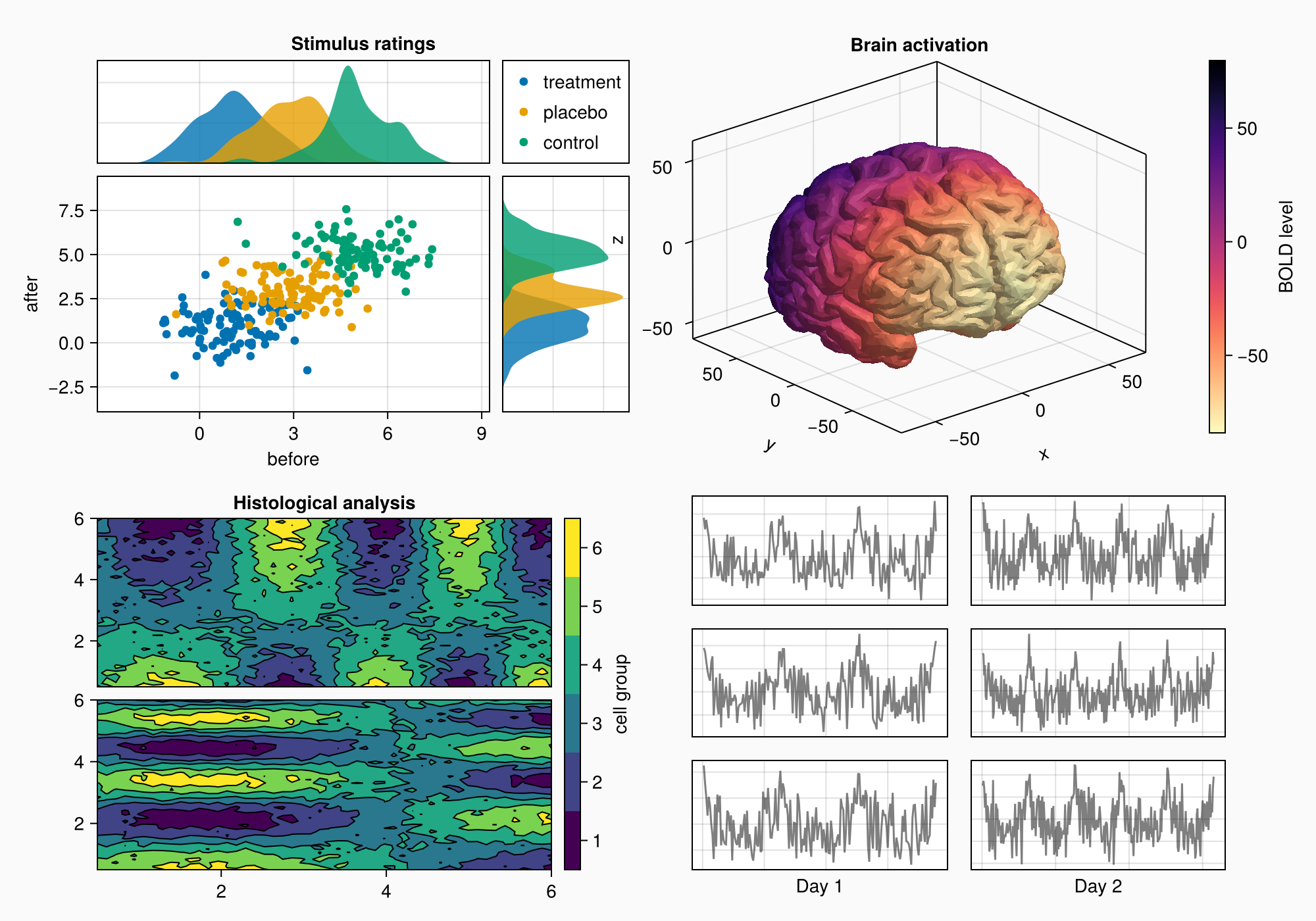
Можно сделать небольшой заголовок для шести осей, поместив Label в верхнем выступе строки 1 и протянув его через оба столбца.
Label(gd[1, :, Top()], "EEG traces", valign = :bottom,
font = :bold,
padding = (0, 0, 5, 0))
f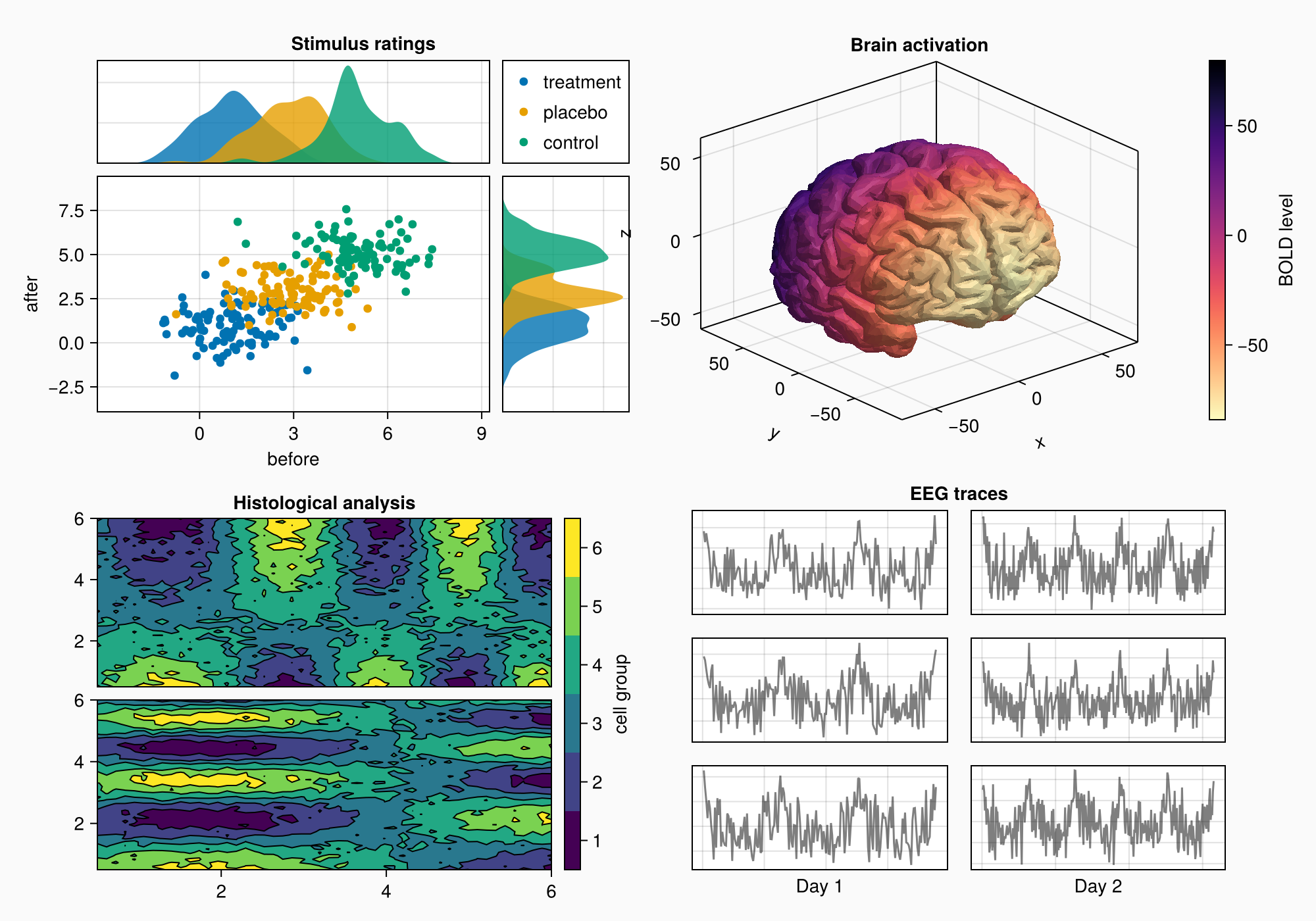
rowgap!(gd, 10)
colgap!(gd, 10)
f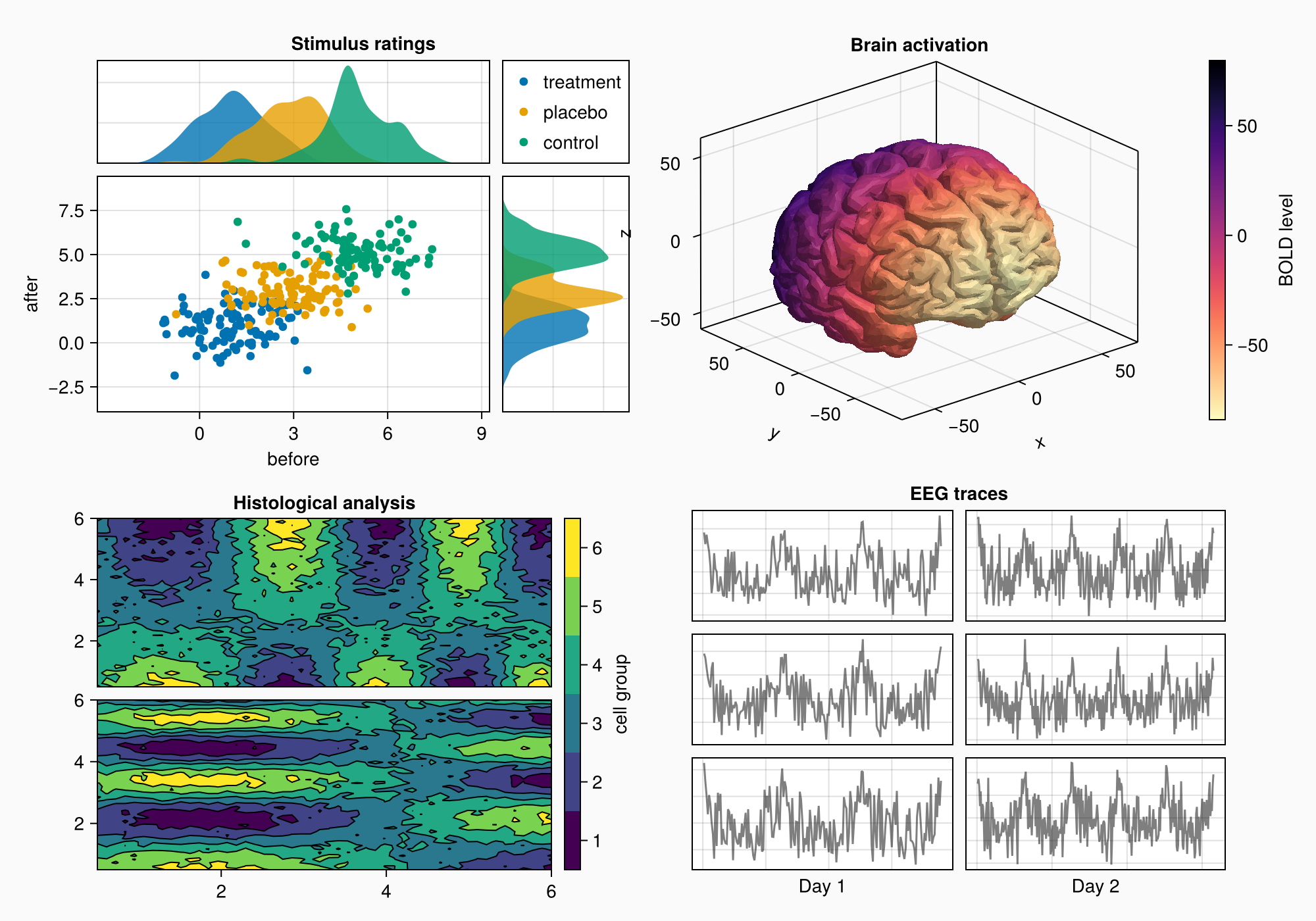
Метки ЭЭГ
Теперь добавим сбоку три прямоугольника с метками внутри. В этом случае мы просто поместим их в другой столбец справа.
for (i, label) in enumerate(["sleep", "awake", "test"])
Box(gd[i, 3], color = :gray90)
Label(gd[i, 3], label, rotation = pi/2, tellheight = false)
end
f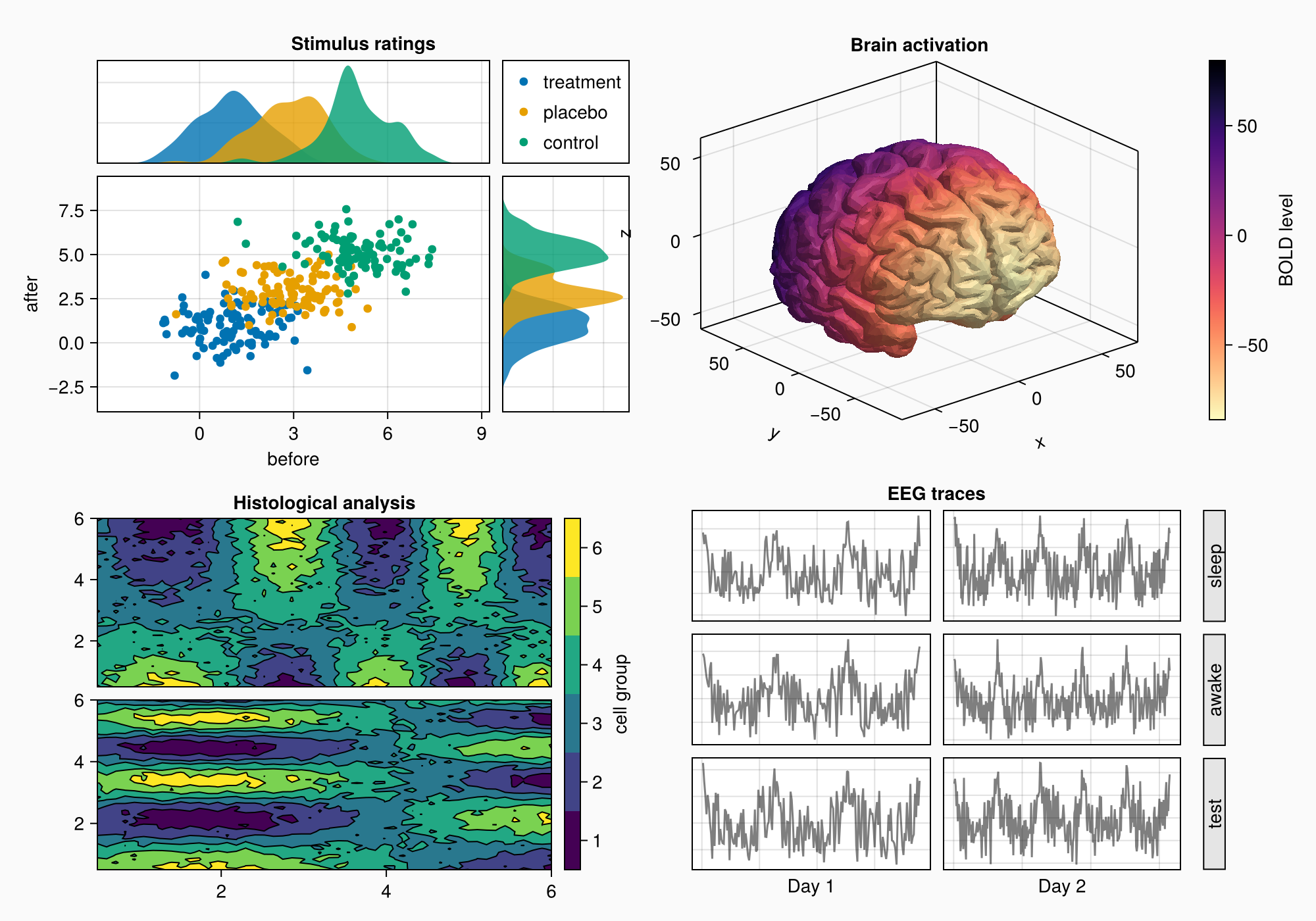
Прямоугольники находятся в правильном положении, но нам все еще нужно убрать промежуток между столбцами.
colgap!(gd, 2, 0)
f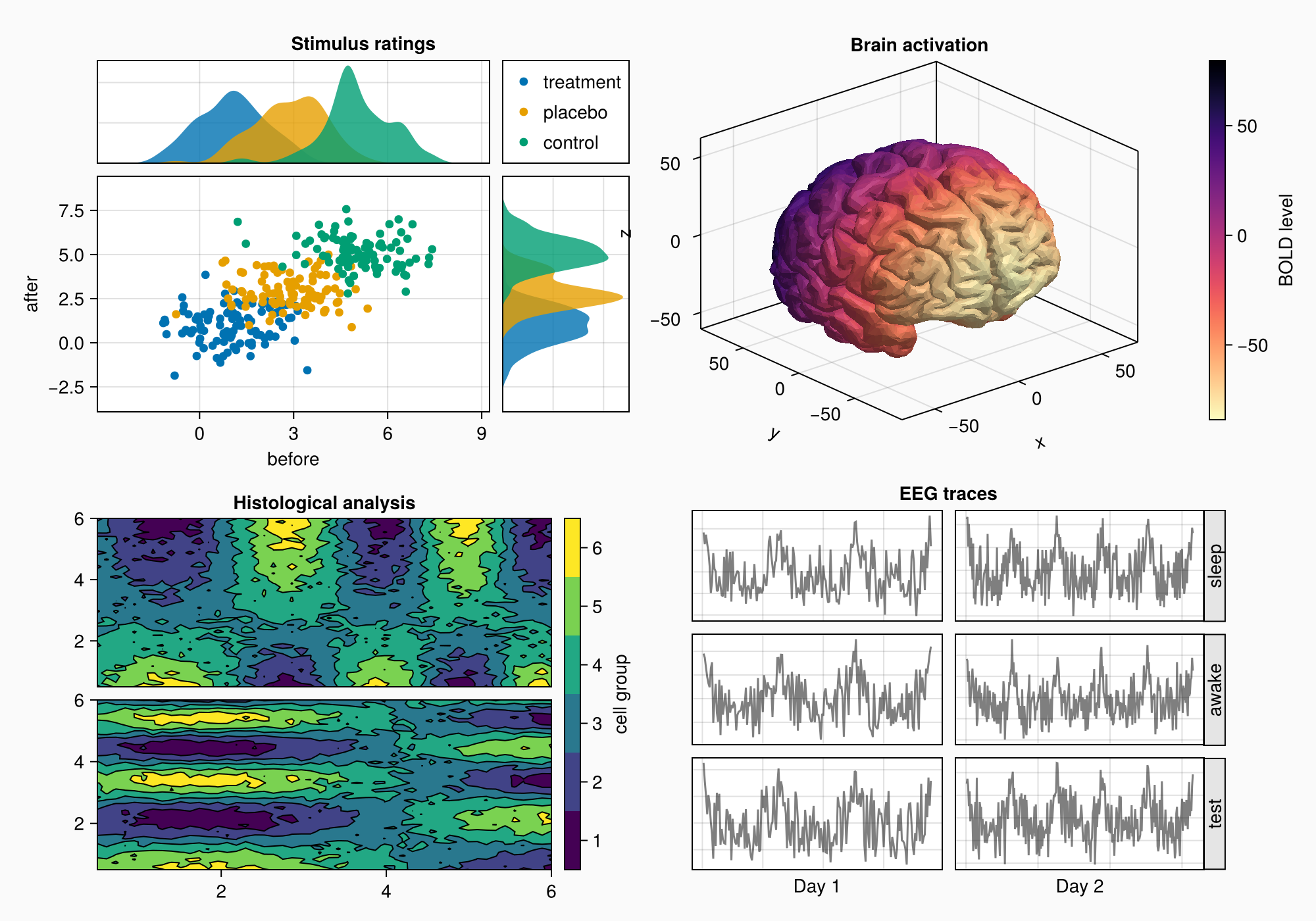
Относительное масштабирование осей
Созданные фиктивные данные ЭЭГ содержат больше точек данных во второй день, чем в первый. Нужно масштабировать оси так, чтобы они обе имели одинаковый уровень масштабирования. Для этого зададим для ширины столбцов режим Auto(x), где x — число, пропорциональное количеству точек данных на оси. Таким образом, они будут иметь одинаковый относительный масштаб.
n_day_1 = length(0:0.1:6pi)
n_day_2 = length(0:0.1:10pi)
colsize!(gd, 1, Auto(n_day_1))
colsize!(gd, 2, Auto(n_day_2))
f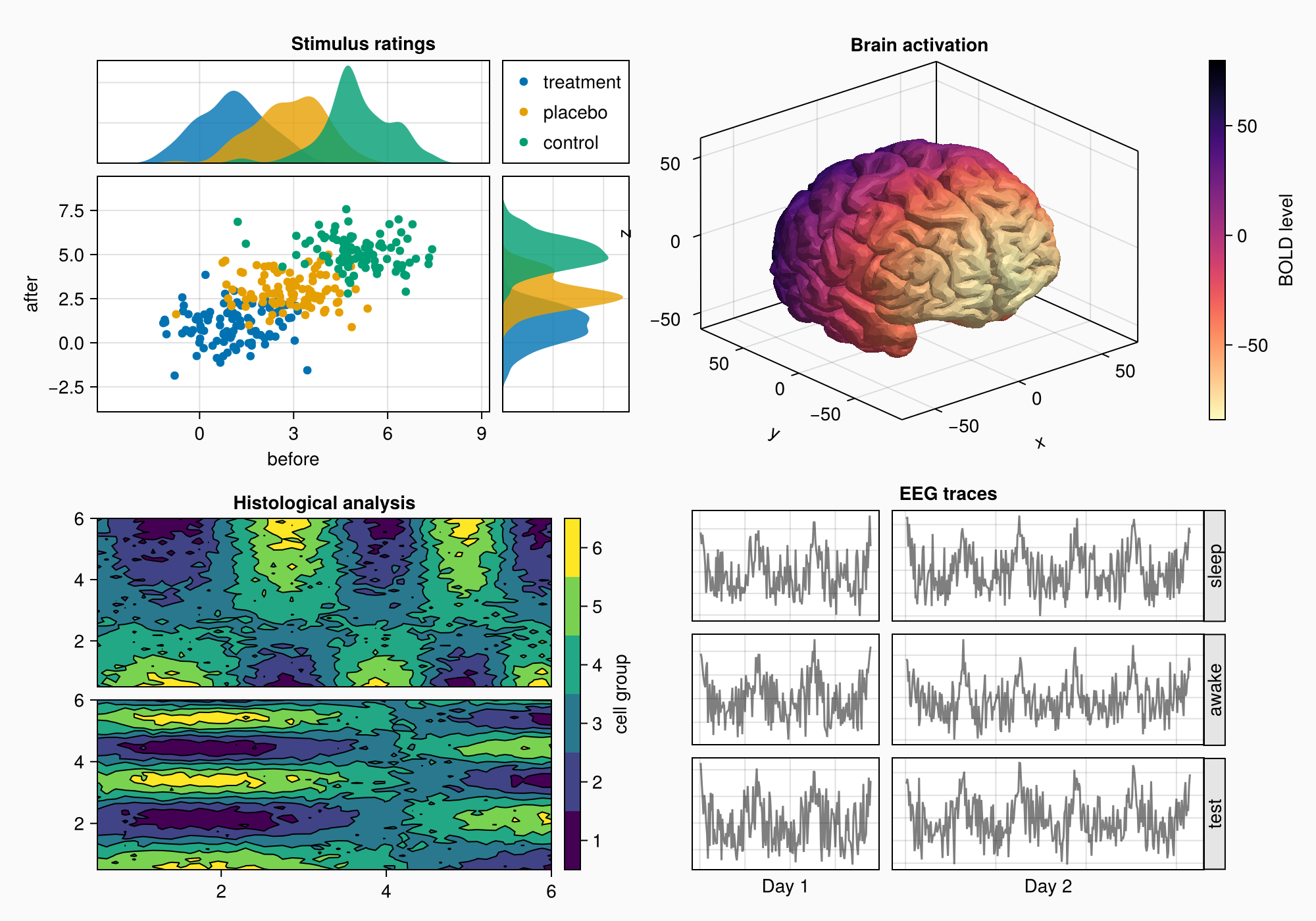
Метки подграфика
Теперь можно добавить метки для подграфика. У нас уже есть четыре объекта GridLayout, которые заключают содержимое каждой панели, поэтому проще всего будет создать Label в левом верхнем выступе этих макетов. При этом все остальные выравнивания останутся нетронутыми, поскольку новые столбцы или строки не создаются. И метки относятся к промежуткам между макетами.
for (label, layout) in zip(["A", "B", "C", "D"], [ga, gb, gc, gd])
Label(layout[1, 1, TopLeft()], label,
fontsize = 26,
font = :bold,
padding = (0, 5, 5, 0),
halign = :right)
end
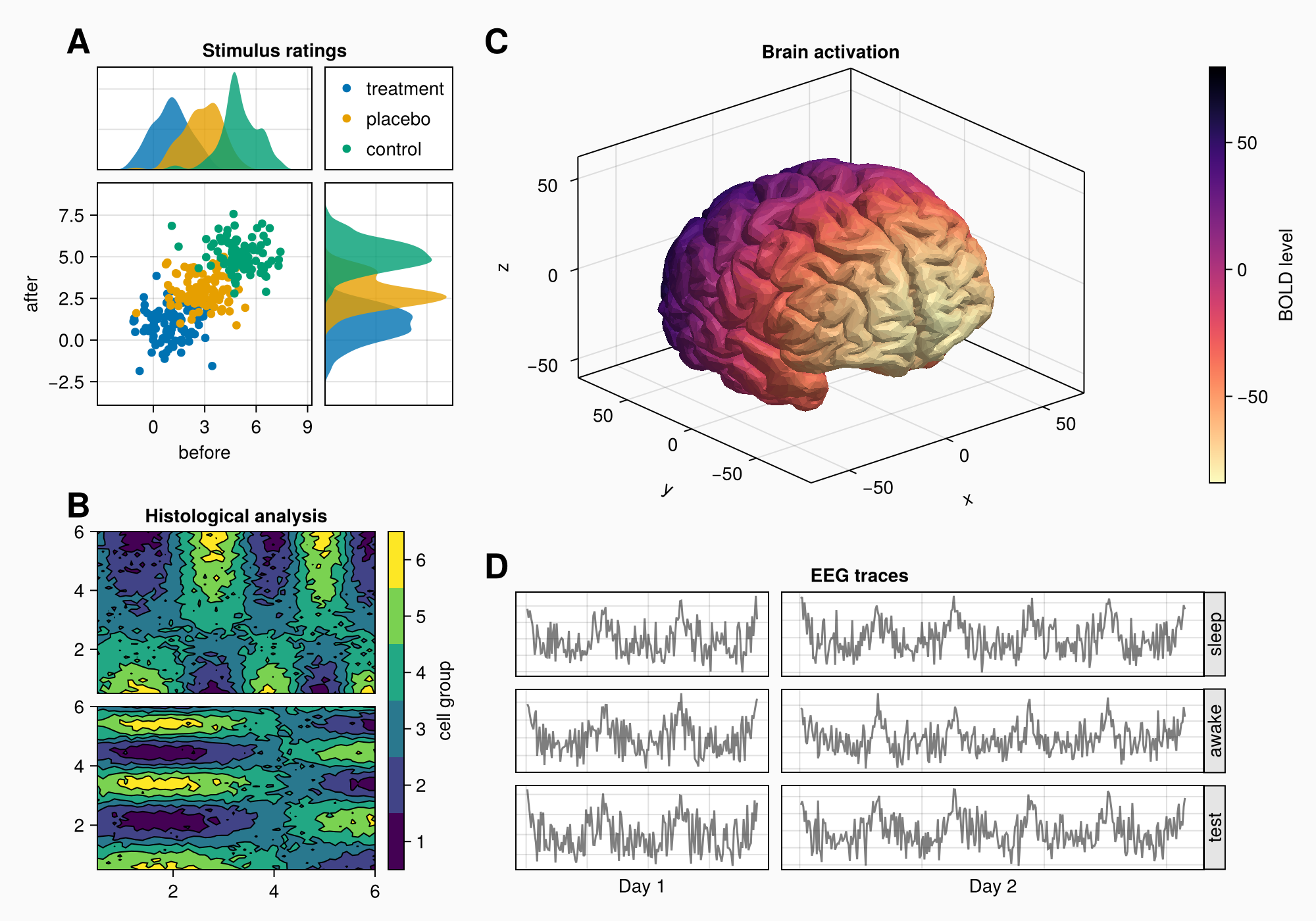
f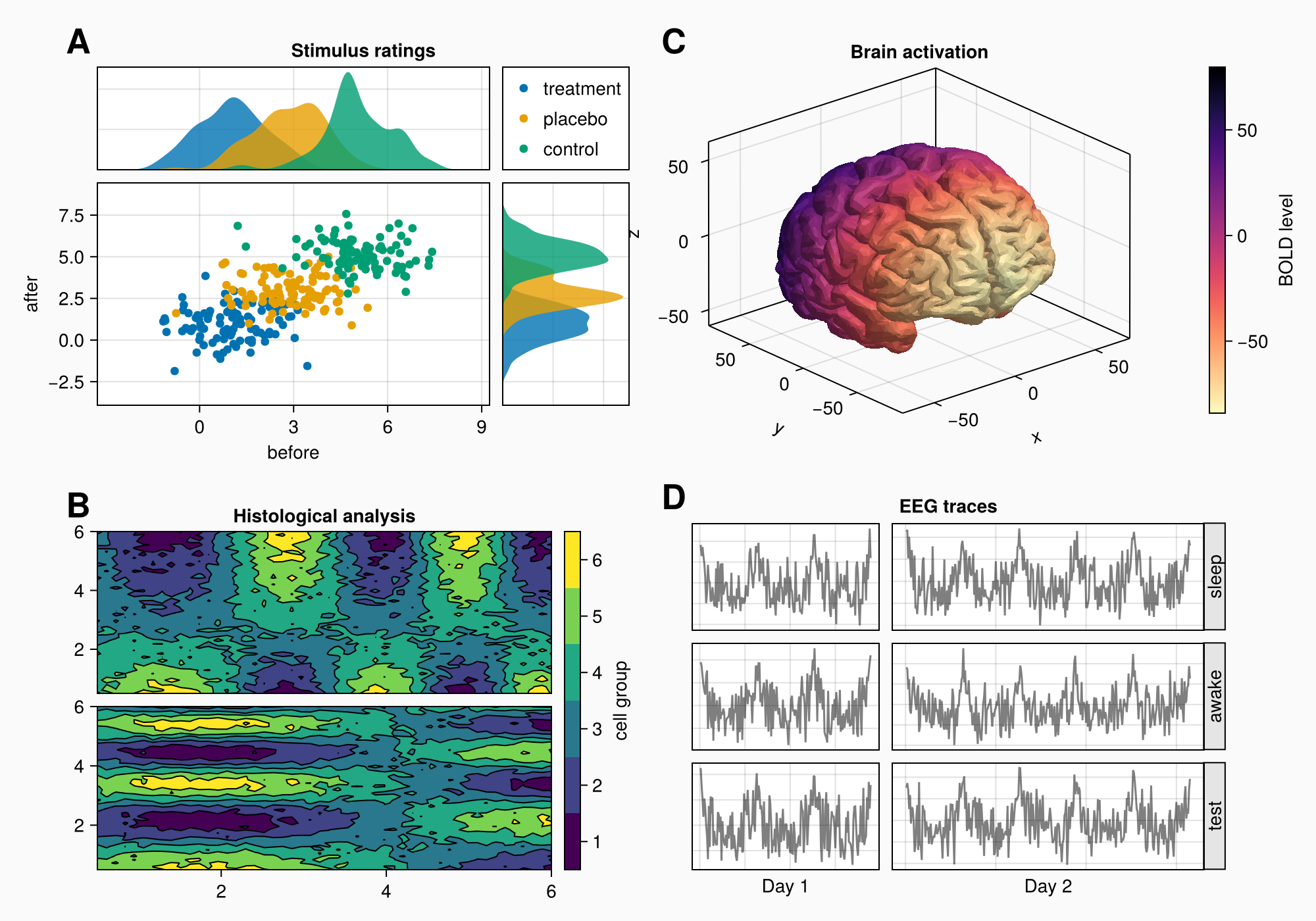
Окончательные настройки
Все выглядит уже неплохо, но первый столбец макета слишком широкий. Чтобы уменьшить ширину столбца, зададим Auto с числом, меньшим 1, например: Так столбец получит меньший вес при распределении значений ширины между всеми столбцами с размерами Auto.
Можно также использовать Relative или Fixed, но они не столь гибкие, если позже планируется добавить что-то еще. Поэтому рекомендуется выбрать Auto.
colsize!(f.layout, 1, Auto(0.5))
f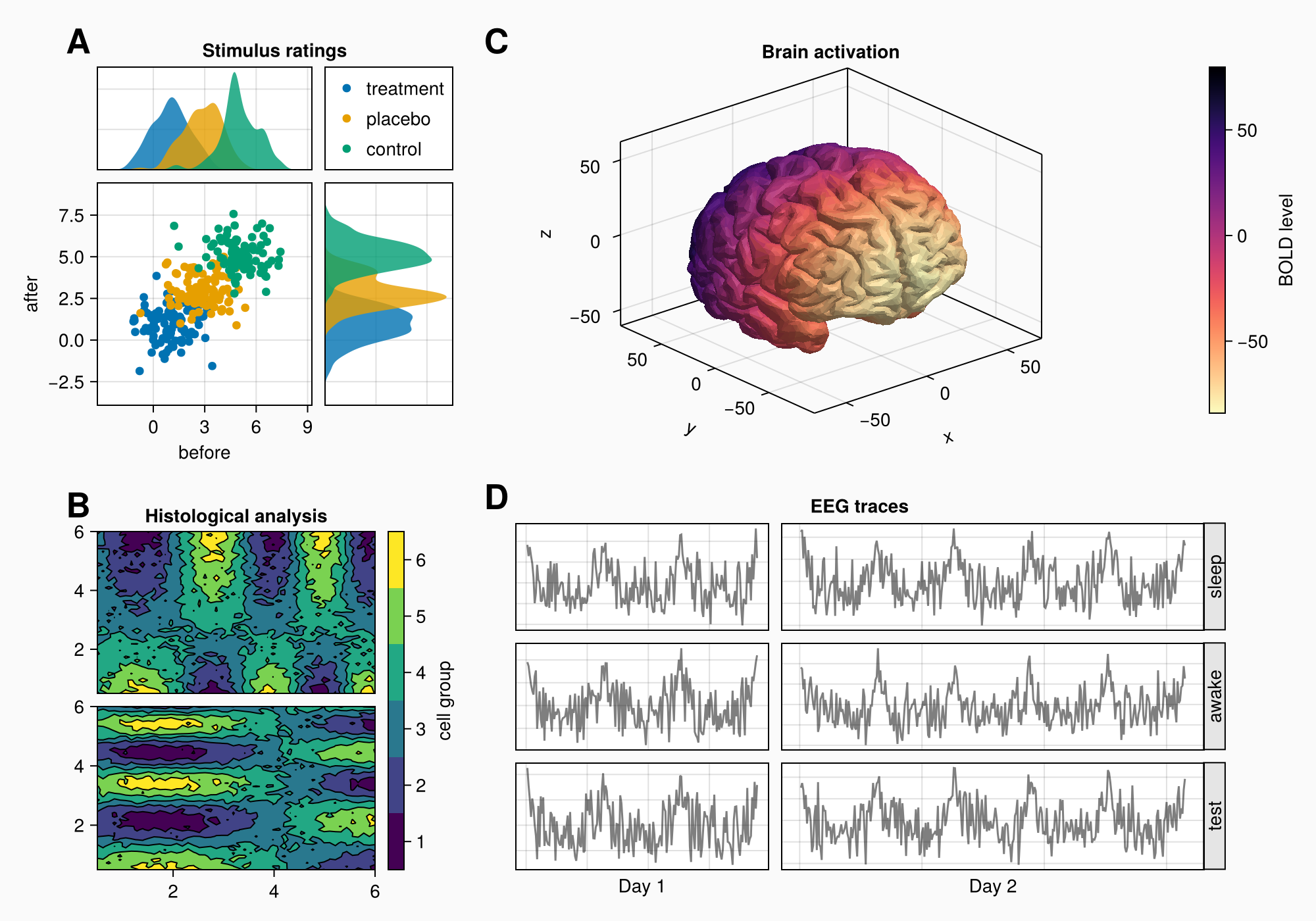
Сейчас записи ЭЭГ находятся на той же высоте, что и ось мозга, поэтому давайте немного увеличим размер строки с макетом панели C, чтобы у нее было больше места.
rowsize!(gcd, 1, Auto(1.5))
f